熱力学パラメータの生成(Parameter generation)¶
ESPEI は熱力学パラメータの自動生成を行うことができます。各エンドメンバーに対して、compound energy formalism (CEF)に基づいて、ギブスエネルギーの 温度依存項のべき級数展開係数 を自動生成します。
また2元系、3元系についてはMuggianu 外挿による Redlich-Kister 相互作用パラメータ を自動生成することができます。
モデル生成については、エンタルピー、エントロピー、および熱容量データ(非平衡化学熱力学データを参照) の線形回帰に基づいており、過剰適合を防ぐため修正された 赤池情報量基準(AICc) が使用されています。
ESPEI で熱力学パラメータの生成を行うには、3種類のファイルが必要です。
相ファイル (phase.json, JSON format)
データファイル (JSON format)
インプットファイル (YAML format)
相ファイル(phase.json)¶
元素と相の情報を記述します。ファイル名は慣例により phases.json が使用されます。
Cr-Ni二元系について、
液相(LIQUID)
BCC相(BCC_A2)
FCC相(FCC_A1)
を定義し、それぞれ1つの副格子をもつ固溶体相を定義するには以下のようにします。
{
"components": ["CR", "NI"],
"phases": {
"LIQUID" : {
"sublattice_model": [["CR", "NI"]],
"sublattice_site_ratios": [1]
},
"BCC_A2": {
"sublattice_model": [["CR", "NI"]],
"sublattice_site_ratios": [1]
},
"FCC_A1": {
"sublattice_model": [["CR", "NI"]],
"sublattice_site_ratios": [1]
}
}
}
components:¶
構成元素の定義。」XX」と」」YY」」が含まれる場合は、」components」: [「XX」, 「YY」],とします。
phases:¶
相の定義。」LIQUID」, 「BCC_A2」, 「FCC_A1」を定義しています。各相において、以下の副格子の定義が必要です。
sublattice_model:¶
副格子の数と、各副格子に含まれる元素。2つの副格子に」XX」と」」YY」」が含まれる場合は、」sublattice_model」: [[「XX」, 「YY」],[「XX」, 「YY」]], のようにします。
sublattice_site_ratios:¶
各副格子の化学量論比。2つの副格子の化学量論比1:2にする場合は、 「sublattice_site_ratios」: [1, 2] のようにします。
詳細については、Phase Models Schema を参照してください。
データファイル (JSON)¶
ESPEIでフィッティングをする際には、以下の3種類のデータを使用することができます。
熱化学データ(非平衡)
熱化学データ(平衡)
相境界データ
パラメータ生成(Parameter generation) では、非平衡状態の熱化学データのみが使用できます。この熱化学データは内部自由度が既知である場合の熱力学量で、安定相、準安定相、不安定相の全てについて使用することができます。
マルコフ連鎖モンテカル法(MCMC) では、すべての種類のデータを使用することができます。熱化学データ(平衡)は、安定相または準安定相の熱力学量が、相境界データは準安定相が安定に、また安定相が準安定になる組成の条件を含みます。
詳細については、ESPEI Dataset Schemaを参照してください。以下の例では、熱化学データ(非平衡)と相境界データのみを使用します。
熱化学データ(非平衡)(Non-equilibrium thermochemical data)¶
論文として発表されている生成エンタルピーのデータをフィッティングに使用する場合には、CR-NI-HM_FORM-FCC_A1-watson1995enthapies.json のように、ファイルの内容が分かりやすいファイル名を使用することが推奨されています。
{
"components": ["CR", "NI"],
"phases": ["FCC_A1"],
"solver": {
"mode": "manual",
"sublattice_site_ratios": [1],
"sublattice_configurations": [ [["CR", "NI"]], [["CR", "NI"]], [["CR", "NI"]], [["CR", "NI"]]],
"sublattice_occupancies": [ [[0.10, 0.90]], [[0.10, 0.90]], [[0.40, 0.60]], [[0.40, 0.60]]]
},
"conditions": {
"P": 101325,
"T": 1583
},
"output": "HM_FORM",
"values": [[[-1100, -1510, 2880, 3280]]],
"reference": "watson1995enthapies",
"comment": "From Table 5. Converted from kJ to J. Two phase data neglected."
}
components:¶
元素を記述します。
phases:¶
相を記述します。
solver:¶
sublattice_site_ratios:副格子の数
sublattice_configurations:副格子を構成する元素
sublattice_occupancies:副格子の組成比をデータ数記述
conditions:¶
P:圧力
T:温度
スカラーまたは 1 次元リストとして記述します。
output:¶
データ(熱力学量)を指定します。現在は CPM、SM、HM、SM_MIX、HM_MIX、HM_FORM がサポートされています。標準状態を変更する機能はまだ実装されていないため、すべての熱力学的量は形成量 (例:HM_FORM または HM_MIX など) である必要があります。
values:¶
指定した圧力、温度、副格子配置に対応する output の値です。3 次元配列データで、配列のサイズは (len(P), len(T), len(subl_config)) です。
reference:¶
comment:¶
また以下の例では、圧力が 1 つ、副格子配置が 1 つ、温度が 12 個あるため、values 配列の形状は (1, 12, 1) です。
{
"reference": "Yi Wang et al 2009",
"components": ["AL", "NI", "VA"],
"phases": ["BCC_B2"],
"solver": {
"mode": "manual",
"sublattice_site_ratios": [0.5, 0.5, 1],
"sublattice_configurations": [["AL", "NI", "VA"]],
"comment": "NiAl sublattice configuration (2SL)"
},
"conditions": {
"P": 101325,
"T": [ 0, 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, 110]
},
"excluded_model_contributions": ["idmix", "mag"],
"output": "CPM_FORM",
"values": [[[ 0 ],
[-0.0173 ],
[-0.01205],
[ 0.12915],
[ 0.24355],
[ 0.13305],
[-0.1617 ],
[-0.51625],
[-0.841 ],
[-1.0975 ],
[-1.28045],
[-1.3997 ]]]
}
excluded_model_contributions:¶
熱力学モデルのフィッティングの対象から外す場合に使用します。これはフィッティング対象のデータに寄与が含まれていない場合に役立ちます。
例えばDFTの形成エネルギーには、理想的な混合エンタルピーは含まれません。また、DFTの形成エネルギーにはCalphad タイプの磁気モデルの寄与は含まれません。ただし実験の形成エネルギーにはこれらの寄与が含まれるため、実験の形成エネルギーを使用する場合には除外してはいけません。
相境界データ(Phase boundary data)¶
平衡状態にある相境界データをフィッティングに用いることができます。相境界データの JSON データセットは、"output": "ZPF" を使用します。値は、特定の温度および圧力条件下で 1 つ以上の相が平衡状態にある相境界に対応します。
{
"components": ["CR", "NI"],
"phases": ["BCC_A2", "FCC_A1"],
"broadcast_conditions": false,
"conditions": {
"T": [1073, 1173, 1273, 1373, 1548],
"P": [101325.0]
},
"output": "ZPF",
"values": [
[["FCC", ["CR"], [0.3866]], ["BCC", ["NI"], [null]]],
[["FCC", ["CR"], [0.3975]], ["BCC", ["NI"], [null]]],
[["FCC", ["CR"], [0.4480]], ["BCC", ["NI"], [null]]],
[["FCC", ["CR"], [0.4643]], ["BCC", ["NI"], [null]]],
[["FCC", ["CR"], [0.4984]], ["BCC", ["NI"], [null]]]
],
"reference": "bechtoldt1961redetermination",
"comment": "Digitized from figure 5, points in figure 4 were too far apart to identify a boundary properly."
}
上の例ではFCC相のCr濃度は数値で指定されていますが、BCC相のNi濃度はnullとなっており不明であることを表しています。これらの組成は、相の内部組成 (全体の組成ではありません) である必要があります。
「相組成」は、二元系状態図の 2 相領域における「タイライン組成」と同じですが、単相平衡や 3 相以上の平衡のようにタイラインの意味があいまいな場合のより一般的な用語です。
相平衡では、1 つ以上の相組成が不明な場合があります。これらの相境界データでは、1 つの相の組成は決定されますが、平衡している他の相の組成は決定されません。これらの場合、相組成は null として指定でき、ESPEI は相組成を推定します。
重要:各相領域には、規定の相組成を持つ相が少なくとも 1 つ必要です。相領域内のすべての相の相組成が null の場合、ターゲット超平面 ((Bocklund 他 2019) の図 1 で説明) は未定義になり、駆動力は計算されません。
重要:c 成分を含むデータセットの場合、各相組成は c-1 成分で指定する必要があります。暗黙の N=1 条件があります。
broadcast_conditions:¶
インプットファイル (YAML)¶
フィッティング手順を記述し、ハイパーパラメータの調整をするYAMLファイルです。詳細については、YAML Input Schemaを参照してください。
[1]:
!cat generate_params_settings.yaml
system:
phase_models: Cr-Ni_phases.json
datasets: input-data
tags:
dft:
excluded_model_contributions: ['idmix', 'mag']
weight: 0.1
nomag:
excluded_model_contributions: ['mag']
estimated-entropy:
excluded_model_contributions: ['idmix', 'mag']
weight: 0.1
generate_parameters:
excess_model: linear
ref_state: SGTE91
output:
verbosity: 1
output_db: generated.tdb
system:¶
フィッティングを行う系の元素、相、データなどを記述します。
generate_parameters:¶
パラメータの選択と単相データへのフィッティングを制御するために使用されます。熱容量や混合エネルギーなどの熱化学データを入力する必要がある場合は、このオプションを使用する必要があります。パラメータ生成では、赤池情報量基準を使用してモデルパラメータを選択し、それを用いてフィッティングを行いデータベースを作成します。
excess_model:¶
過剰混合パラメータに使用するモデルを指定します。現在は線形モデルのみがサポートされており、指数モデルおよびカスタムモデルのサポートが計画されています。
ref_state:¶
純元素と格子安定性に使用される参照状態。現在はSGTE91とSR2016(一部の要素)のみがサポートされています。カスタム参照状態をサポートするための拡張が計画されています。
output:¶
verbosity:¶
ログレベルを制御します。ほとんどの場合、Info または Trace を使用します。Warningは静かで、Debugは大量の情報を提供し、計算エラーの修正や重みの調整に役立ちます。
Value |
Log Level |
|---|---|
0 |
Warning |
1 |
Info |
2 |
Trace |
3 |
Debug |
パラメータ生成¶
ESPEIを実行する前にデータセットを確認することが推奨されます。ESPEIには、データセット内の問題を検出して報告するためのツールがあります。このツールはデータセットをロードすると自動的に実行されますが、最初のエラーが発生したところで終了します。そこで以下のコマンドを実行してください。
[1]:
!espei --check-datasets input-data
/Users/iikubo/miniconda3/envs/pycalphad/lib/python3.9/site-packages/pydantic/_migration.py:281: UserWarning: `pydantic:PyObject` has been moved to `pydantic.types:ImportString`.
warnings.warn(f'`{import_path}` has been moved to `{new_location}`.')
これを実行すると、すべてのデータセットが一度にチェックされ、問題点が報告されます。エラーが発生した場合には、そのリストが報告されます。エラーには主に2種類あり、JSONError(データセットスキーマのJSONセクションを参照)とDatasetError(データセットの妥当性(一致条件や値の形状など)に関連するエラー)です。
以下のプログラムを実行すると、状態図を表示して実験データと比較することができます。
[8]:
import yaml
from espei import run_espei
from pycalphad import Database, binplot, equilibrium, variables as v
with open('generate_params_settings.yaml') as fp:
generate_params_settings = yaml.safe_load(fp)
dbf = run_espei(generate_params_settings)
comps = ['CR', 'NI']
phases = ['FCC_A1', 'BCC_A2', 'LIQUID']
conds = {v.N: 1.0, v.P: 101325, v.T: (300, 2300, 20), v.X('NI'): (0, 1, 0.02)}
binplot(dbf, comps, phases, conds)
INFO:espei.espei_script - espei version 0.0.0
INFO:espei.espei_script - pycalphad version 0.10.2
INFO:espei.espei_script - dask version 2023.6.0
INFO:espei.espei_script - distributed version 2023.6.0
INFO:espei.espei_script - symengine version 0.9.2
INFO:espei.espei_script - emcee version 2.2.1
INFO:espei.espei_script - If you use ESPEI for work presented in a publication, we ask that you cite the following paper:
B. Bocklund, R. Otis, A. Egorov, A. Obaied, I. Roslyakova, Z.-K. Liu, ESPEI for efficient thermodynamic database development, modification, and uncertainty quantification: application to Cu-Mg, MRS Commun. (2019) 1-10. doi:10.1557/mrc.2019.59.
INFO:espei.paramselect - Generating parameters.
INFO:espei.paramselect - FITTING: BCC_A2
INFO:espei.paramselect - FITTING: FCC_A1
INFO:espei.paramselect - FITTING: LIQUID
INFO:espei.paramselect - Finished generating parameters.
[8]:
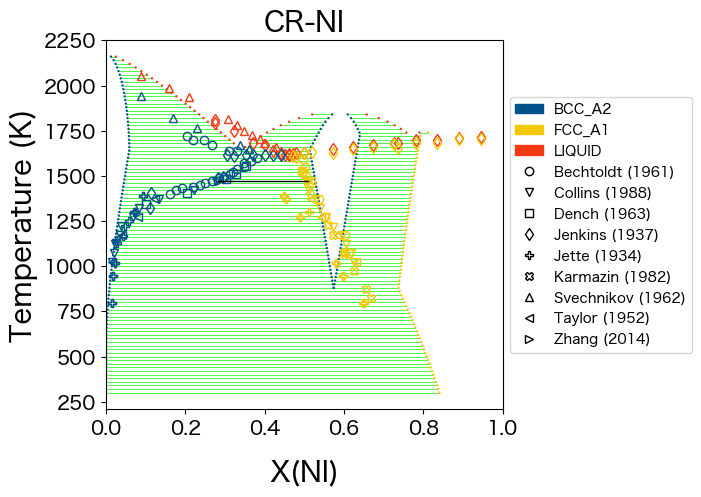
<Axes: title={'center': 'CR-NI'}, xlabel='X(NI)', ylabel='Temperature (K)'>
ギブスエネルギーの導関数に対する初期パラメータが得られたので、これを実験状態図と比較します。すべてのデータはinput-dataディレクトリ以下にJSONファイルとして保存されています。それを datasets としてロードします。
[10]:
!ls input-data
activity zpf
non-equilibrium-thermochemical
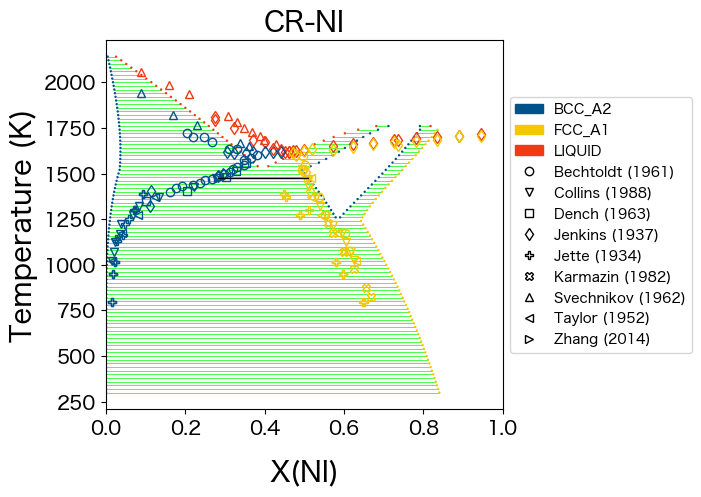
次にこれらの新しいデータセットを同じ軸上に使用して二元状態図をプロットします。
[13]:
from espei.plot import dataplot
from espei.datasets import recursive_glob, load_datasets
# load our JSON datasets into an in-memory database
datasets = load_datasets(recursive_glob('input-data', '*.json'))
# plot the binary phase diagram, saving the output matplotlib axes as a variable
ax = binplot(dbf, comps, phases, conds)
# plot the phase boundary data as marked points
dataplot(comps, phases, conds, datasets, ax=ax)
[13]:
<Axes: title={'center': 'CR-NI'}, xlabel='X(NI)', ylabel='Temperature (K)'>
ギブスエネルギーの確認¶
pycalphadを使用してギブスエネルギーを図示し、BCC相が安定する原因について理解します。
[19]:
from pycalphad import calculate
import matplotlib.pyplot as plt
calc_res = calculate(dbf, comps, phases, T=1500, N=1, P=101325)
for phase_name in phases:
mask = calc_res.Phase == phase_name
plt.scatter(calc_res.X.sel(component='NI'), calc_res.GM.where(mask), s=2, label=phase_name)
plt.legend()
plt.xlabel("X(NI)")
plt.ylabel("GM (J/mol-atom)")
plt.title("Cr-Ni Energy Surface")
/Users/iikubo/miniconda3/envs/pycalphad/lib/python3.9/site-packages/pycalphad/core/utils.py:54: RuntimeWarning: invalid value encountered in divide
pts[:, cur_idx:end_idx] /= pts[:, cur_idx:end_idx].sum(axis=1)[:, None]
[19]:
Text(0.5, 1.0, 'Cr-Ni Energy Surface')
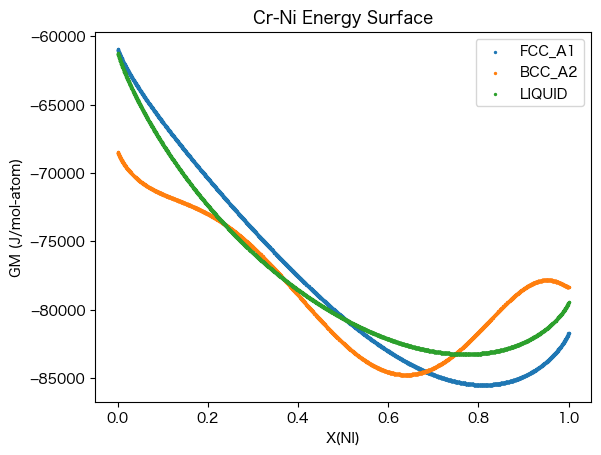
BCC相は奇妙な形をしているように見えます。パラメーターを調査すると、根本的な問題が明らかになる可能性があります。
ESPEI では、相互作用パラメーターを最適化する目的で plot_interaction が使用できます。
[21]:
from espei.plot import plot_interaction
plot_interaction(dbf, comps, 'BCC_A2', (('CR', 'NI'),), 'HM_MIX', datasets=datasets, symmetry=None)
#plot_interaction(dbf, comps, 'FCC_A1', (('CR', 'NI'),), 'HM_MIX', datasets=datasets, symmetry=None)
#plot_interaction(dbf, comps, 'LIQUID', (('CR', 'NI'),), 'HM_MIX', datasets=datasets, symmetry=None)
[21]:
<Axes: xlabel='CR to NI', ylabel='Enthalpy of Mixing (J/mol-atom)'>
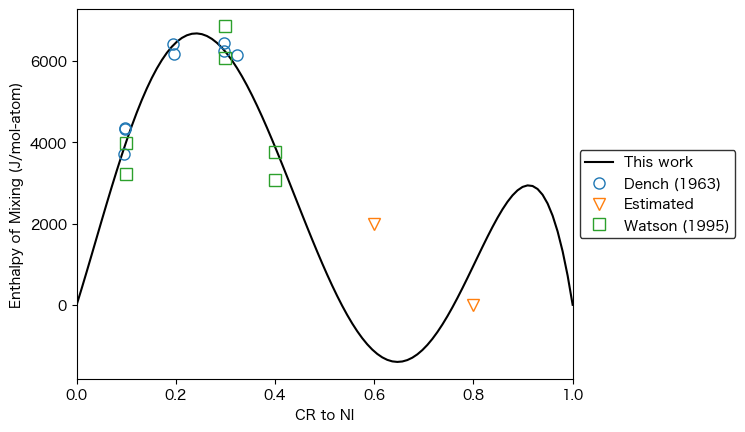
より定量的にするために、パラメータを検索して精密化した過剰パラメータを調べることができます。
[18]:
import tinydb
dbf._parameters.search(
(tinydb.where('phase_name') == 'BCC_A2') & # LIQUID phase parameters
(tinydb.where('parameter_type') == 'L') # Parameters of type L (i.e. excess parameters)
)
[18]:
[{'phase_name': 'BCC_A2',
'constituent_array': ((Species('CR', 'CR1'), Species('NI', 'NI1')),),
'parameter_type': 'L',
'parameter_order': 0,
'parameter': VV0006 + T*VV0005,
'diffusing_species': None,
'reference': None},
{'phase_name': 'BCC_A2',
'constituent_array': ((Species('CR', 'CR1'), Species('NI', 'NI1')),),
'parameter_type': 'L',
'parameter_order': 1,
'parameter': VV0004 + T*VV0003,
'diffusing_species': None,
'reference': None},
{'phase_name': 'BCC_A2',
'constituent_array': ((Species('CR', 'CR1'), Species('NI', 'NI1')),),
'parameter_type': 'L',
'parameter_order': 2,
'parameter': VV0002 + T*VV0001,
'diffusing_species': None,
'reference': None},
{'phase_name': 'BCC_A2',
'constituent_array': ((Species('CR', 'CR1'), Species('NI', 'NI1')),),
'parameter_type': 'L',
'parameter_order': 3,
'parameter': VV0000,
'diffusing_species': None,
'reference': None}]
各過剰パラメータの数式は、parameter キーで確認することができます。最初のparameter は、
$ \mathrm{VV0005} \(と\) \mathrm{VV0006} \(はESPEIによって生成されるパラメータです。ここで`dbf.symbols`を使うと、データベース内パラメータと値をマッピングして\) :nbsphinx-math:`mathrm{VV0006}`$の値を参照できます。
[13]:
print(f"L0-BCC_A2 = {dbf.symbols['VV0006']} + {dbf.symbols['VV0005']}*T")
L0-BCC_A2 = 3527.86 + -3.45952*T
パラメータ選択の調整¶
BCC_A2のパラメータ生成を再度見てみると、修正されたAkiake情報量基準に基づく適合度とパラメータ数の間の最適なトレードオフは、4つの過剰パラメータ(\({}^{0}L\)、\({}^{1}L\)、\({}^{2}L\)、\({}^{3}L\))を選択することであったことがわかります。今回この選択は、目視による判断では、間違っていた可能性があります。
ESPEIには、自動パラメータ生成を調整する方法があります。その一つが、aicc_penalty_factorオプション(詳細はこちら)です。これを利用して、パラメータ数が増える際のペナルティを増やすことができます。
デフォルトでは、すべての相、およびデータタイプにおいて aicc_penalty_factor = 1.0 です。
aicc_penalty_factor > 1 を指定すると、パラメータ数の増加に対するペナルティが増加します。BCC相と液相では、以下のようにペナルティ係数を増やすことができます (generate_params_settings_aicc_penalty.yaml)。
system:
phase_models: Cr-Ni_phases.json
datasets: input-data
tags:
dft:
excluded_model_contributions: ['idmix', 'mag']
weight: 0.1
nomag:
excluded_model_contributions: ['mag']
estimated-entropy:
excluded_model_contributions: ['idmix', 'mag']
weight: 0.1
generate_parameters:
excess_model: linear
ref_state: SGTE91
aicc_penalty_factor:
BCC_A2:
HM: 2.0
SM: 2.0
LIQUID:
HM: 10
SM: 10
output:
verbosity: 1
output_db: generated-aicc.tdb
次に、新しいデータベースを適合させます。
[23]:
with open('generate_params_settings-aicc_penalty.yaml') as fp:
generate_params_settings = yaml.safe_load(fp)
dbf_aicc_penalty = run_espei(generate_params_settings)
INFO:espei.espei_script - espei version 0.0.0
INFO:espei.espei_script - pycalphad version 0.10.2
INFO:espei.espei_script - dask version 2023.6.0
INFO:espei.espei_script - distributed version 2023.6.0
INFO:espei.espei_script - symengine version 0.9.2
INFO:espei.espei_script - emcee version 2.2.1
INFO:espei.espei_script - If you use ESPEI for work presented in a publication, we ask that you cite the following paper:
B. Bocklund, R. Otis, A. Egorov, A. Obaied, I. Roslyakova, Z.-K. Liu, ESPEI for efficient thermodynamic database development, modification, and uncertainty quantification: application to Cu-Mg, MRS Commun. (2019) 1-10. doi:10.1557/mrc.2019.59.
INFO:espei.paramselect - Generating parameters.
INFO:espei.paramselect - FITTING: BCC_A2
INFO:espei.paramselect - FITTING: FCC_A1
INFO:espei.paramselect - FITTING: LIQUID
INFO:espei.paramselect - Finished generating parameters.
BCC_A2 相の新しいパラメータを検証します。
[24]:
dbf_aicc_penalty._parameters.search(
(tinydb.where('phase_name') == 'BCC_A2') & # LIQUID phase parameters
(tinydb.where('parameter_type') == 'L') # Parameters of type L (i.e. excess parameters)
)
[24]:
[{'phase_name': 'BCC_A2',
'constituent_array': ((Species('CR', 'CR1'), Species('NI', 'NI1')),),
'parameter_type': 'L',
'parameter_order': 0,
'parameter': VV0003 + T*VV0002,
'diffusing_species': None,
'reference': None},
{'phase_name': 'BCC_A2',
'constituent_array': ((Species('CR', 'CR1'), Species('NI', 'NI1')),),
'parameter_type': 'L',
'parameter_order': 1,
'parameter': VV0001 + T*VV0000,
'diffusing_species': None,
'reference': None}]
今回は2つの過剰パラメータ(\({}^{0}L\)、\({}^{1}L\))を選択していることが分かります。
[25]:
plot_interaction(dbf_aicc_penalty, comps, 'BCC_A2', (('CR', 'NI'),), 'HM_MIX', datasets=datasets, symmetry=None)
[25]:
<Axes: xlabel='CR to NI', ylabel='Enthalpy of Mixing (J/mol-atom)'>
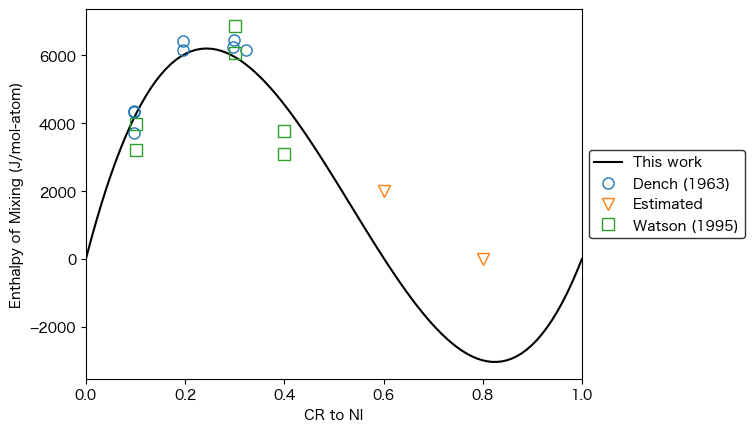
[26]:
calc_res = calculate(dbf_aicc_penalty, comps, phases, T=1500, N=1, P=101325)
for phase_name in phases:
mask = calc_res.Phase == phase_name
plt.scatter(calc_res.X.sel(component='NI'), calc_res.GM.where(mask), s=2, label=phase_name)
plt.legend()
plt.xlabel("X(NI)")
plt.ylabel("GM (J/mol-atom)")
plt.title("Cr-Ni Energy Surface")
/Users/iikubo/miniconda3/envs/pycalphad/lib/python3.9/site-packages/pycalphad/core/utils.py:54: RuntimeWarning: invalid value encountered in divide
pts[:, cur_idx:end_idx] /= pts[:, cur_idx:end_idx].sum(axis=1)[:, None]
[26]:
Text(0.5, 1.0, 'Cr-Ni Energy Surface')
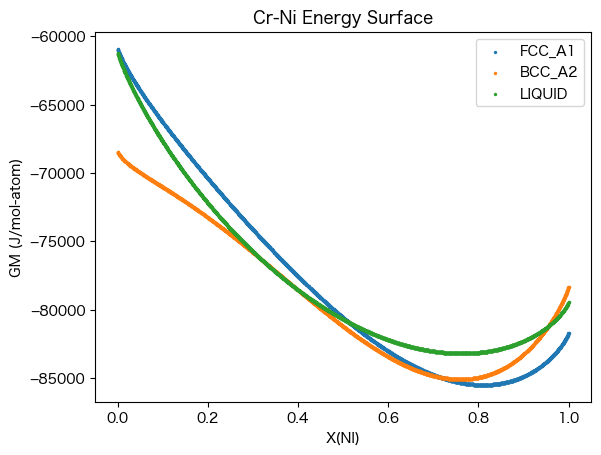
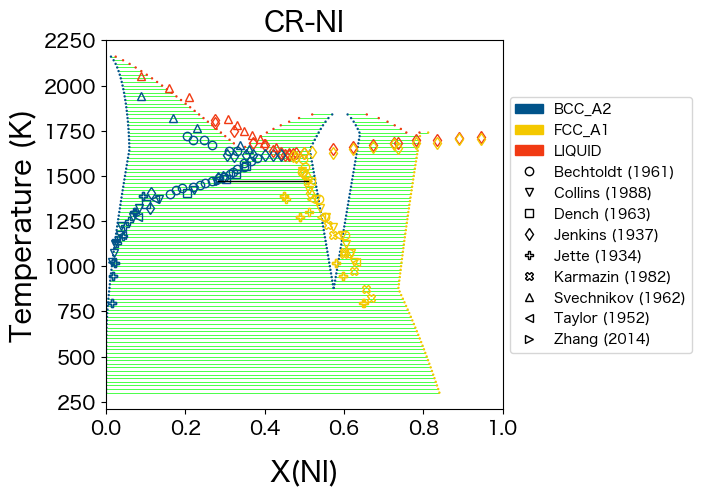
dbfとdbf_aicc_penaltyの結果を比較します。
[27]:
# Original database with four BCC_A2 excess parameters
ax = binplot(dbf, comps, phases, conds)
dataplot(comps, phases, conds, datasets, ax=ax)
# New database with two excess parameters
ax = binplot(dbf_aicc_penalty, comps, phases, conds)
dataplot(comps, phases, conds, datasets, ax=ax)
[27]:
<Axes: title={'center': 'CR-NI'}, xlabel='X(NI)', ylabel='Temperature (K)'>